Forschung an Proteinstrukturen: Die Türsteher der Kernporen
Ein Mandala? Nein, eine kleine medizinische Sensation. Ein Hamburger Team hat es geschafft, den Kernporenkomplex zu visualisieren.
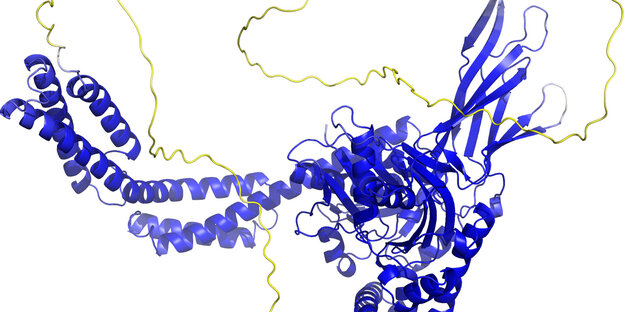
So sieht das Rückgrat der Proteinstruktur aus Foto: AlphaFold Protein Structure Database/dpa
HAMBURG taz | Lila, gelbe, blaue und knallgrüne Fetzen wuchern im Kreis. Was aussieht wie ein buntes Mandala, ist in Wahrheit revolutionär: Endlich gibt es eine strukturelle Abbildung des Kernporenkomplexes.
Wie bitte? Kernporenkomplex? Bei wem der letzte Biologie-Unterricht auch schon ein Weilchen her ist, der fühle sich nicht direkt entmutigt. Eine kleine Auffrischung: Der menschliche Körper besteht aus Milliarden von Zellen, in fast allen befindet sich ein Zellkern. In ihm lagern die Erbinformationen, besser bekannt als DNA. Der Zellkern ist umhüllt von einer Membran – und hier kommen die Kernporen ins Spiel. Sie sind kleine Öffnungen in der Membran.
Alles, was in den Zellkern ein- oder von ihm austreten will, muss es an den Kernporen vorbei schaffen. Wäre der Zellkern ein Club, wären die Kernporen die Türsteher. Und weil es nicht nur einen Türsteher gibt, sondern Tausende in Form verschiedenster Proteine, heißt das Ganze Kernporenkomplex. So, Biostunde beendet. Versprochen.
Um besser zu verstehen, wie die Kernporen-Türsteher ihre Arbeit machen, muss man ihre Struktur genauer kennen. Warum das wichtig ist? Einige Viren, zum Beispiel der Aidserreger HIV, manipulieren die Kernporen zu ihrem Vorteil. Wenn man die Struktur des Kernporenkomplexes besser verstehen lernt, könnte das zukünftig der Bekämpfung feindlicher Viren durch entsprechende Medikamente dienen.Das mit dem Verstehen ist aber nicht ganz so einfach. Bisher war es unmöglich, eine derart komplexe Struktur im Detail dreidimensional zu visualisieren. Jetzt ist es möglich.
Künstliche Intelligenz schafft 3D-Bild
Der in Hamburg forschende Strukturbiologe Dr. Jan Kosinski vom Europäischen Molekularbiologischen Labor (EMBL) hat das zusammen mit seinem Kollegen Dr. Martin Beck vom Max Planck Institut für Biophysik und deren Teams geschafft. Das EMBL und die Londoner Firma DeepMind arbeiten zusammen daran, diese Proteinstrukturen zu veranschaulichen. DeepMind entwickelte die dazu erforderliche Software AlphaFold. Die Software arbeitet mit künstlicher Intelligenz und ist ein wahrer Rechenmeister. Zuerst wurde AlphaFold mit über 100.000 bekannten Datensätzen von Proteinstrukturen trainiert, dann erkannte die KI darin Muster.
Und jetzt? AlphaFold kann inzwischen erstaunlich genau die dreidimensionale Gestalt eines Proteins auf Basis des genetischen Codes voraussagen. Wer hätte gedacht, dass so viel Rechenleistung hinter einem bunten Mandala stecken kann?